
Niet wikkelen, maar vouwen: ook bacteriën organiseren hun DNA (maar dan net even anders)
Sommige bacteriën blijken net als wij eiwitten te hebben die het DNA in hun cellen organiseren. Die doen dat alleen net even anders. Dat blijkt uit nieuw onderzoek van bio-chemici van het Leiden Institute of Chemistry en het Max Planck Institute for Biology. De ontdekking helpt ons beter begrijpen hoe bacteriën hun DNA organiseren en biedt nieuwe inzichten in de evolutie van dit soort eiwitten.
Dat klopt volgens mij niet helemaal, dacht professor Remus Dame toen hij de publicatie van een groep collega-onderzoekers las. ‘Ze hadden eenzelfde eiwitstructuur gevonden als mijn collega's Birte Hernandeze Alvarez en Vikram Alva van het Max Planck Institute for Biology in Tübingen, Duitsland. Zij ontdekten het eiwit een paar jaar geleden. Alleen de manier waarop dit eiwit volgens hen aan het DNA bond, vond ik heel onlogisch.’
DNA veilig en netjes bewaren
Dame en zijn collega’s bestudeerden een speciaal soort eiwit: histonen. Histonen spelen een belangrijke rol in de organisatie van DNA in de cellen van eukaryoten (cellen met celkern) en archaea (eencelligen zonder celkern, zie kader). Dame: ‘DNA-moleculen zijn zeer lang, en bevatten cruciale informatie voor de cel. Om het DNA veilig en compact te bewaren, wikkelt een cel de DNA-strengen strak in elkaar. Dat gebeurt rondom “bolletjes” die gemaakt zijn van histonen.’ Door het DNA op een specifieke manier rondom deze histonbolletjes te draaien, kan de cel ook regelen welke genen wel of niet afleesbaar zijn en dus tot expressie komen.
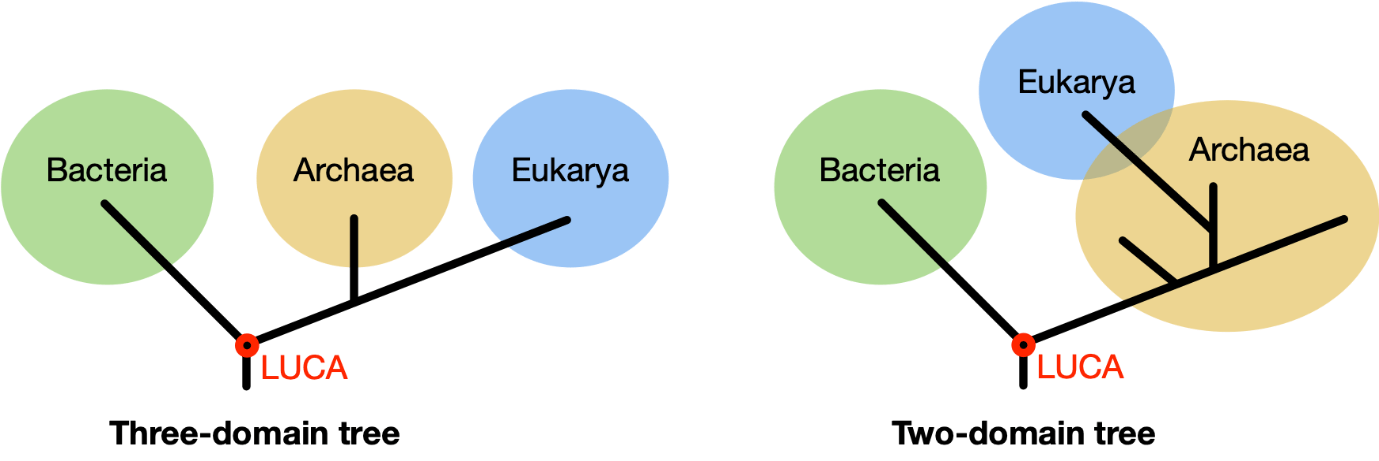
De domeinen van het leven
De meest basale organismen op aarde zijn eencellig en hebben geen celkern. Pas rond 1970 werd ontdekt dat deze organismen twee verschillende groepen vormen, namelijk bacteriën en archaea. Oppervlakkig gezien lijken ze op elkaar, maar hun biochemie is fundamenteel anders, en daarom beschouwen we ze als twee aparte domeinen van het leven. Oorspronkelijk waren organismen met cellen die een kern hebben, de eukaryoten, waar de mens onder valt, ingedeeld in een derde domein. Volgens recente inzichten zijn er echter maar twee domeinen van het leven, en behoren eukaryoten tot de archaea.
'Precies het tegenovergestelde van wat je zou verwachten'
Voor het eerst beschreven zowel de andere onderzoeksgroep als de collega’s van het Max Planck-instituut nu ook een histon-eiwit in een bacterie. ‘Dat is nog niet eerder gedaan’ aldus Dame. ‘De DNA-code en de structuur leken op een simpele versie van ‘onze’ (menselijke) histonen. De vraag was alleen: hebben ze ook dezelfde functie?’ Volgens de concurrerende groep niet, zij beschreven een eiwit dat om het bacterieel DNA gaat zitten en het vervolgens uitrekt. Dame: ‘Precies het tegenovergestelde van wat je zou verwachten. Daarom heb ik contact gezocht met onze collega’s in Duitsland. Yimin Hu, promovenda bij het Max Planck Institute, heeft de structuur van het eiwit-DNA-complex opgelost, en wij wisten hoe we de functie van het eiwit konden bepalen. En toen kwam alles heel mooi samen.’
'Het maakt het DNA juist compact'
Het team van Dame voerde uitgebreide biochemische analyses en ‘single molecule’-experimenten uit. Daarbij bestudeer je individuele moleculen, in plaats van grote aantallen tegelijk. Dame: ‘Zo konden we laten zien dat dit eiwit wel precies doet wat je zou verwachten: het bindt op een heel andere manier aan het bacteriële DNA en maakt het juist compact.’

Niet wikkelen maar vouwen
Maar dat gebeurt wel op een andere manier dan bij andere levensvormen. In eukaryoten vormen histonen structuren die bestaan uit acht eenheden. Samen vormen die een eiwitbol waar het DNA omheen gewikkeld is. In archaea gebeurt dit ook, maar hier is het aantal eenheden oneindig groot. Zo ontstaan staafvormige structuren. In bacteriën blijkt het weer heel anders te zijn: daar vormen de eiwitten twee-eenheden die het DNA niet wikkelen, maar buigen om het compact te maken.
Het onzichtbare zichtbaar maken
DNA is te klein om met het blote oog te zien. Dus hoe onderzoek je het effect van een eiwit op dat DNA? Daarvoor bedachten de onderzoekers een simpele, maar ingenieuze methode. Hierbij voegen ze histon-eiwitten toe aan individuele strengen DNA en observeren wat er gebeurt. De opstelling is vrij eenvoudig: de DNA-streng is aan één kant vastgezet op een glazen plaat, terwijl de andere kant los in het water hangt, als een omgekeerde slinger. Aan dit losse uiteinde komt een plastic bolletje, dat je wél kunt zien, met behulp van een simpele microscoop.

Omdat de bolletjes aan de onderkant vastzitten, is de straal waarbinnen ze kunnen bewegen beperkt. Vervolgens voeg je een eiwit toe. Promovendus Samuel Schwab legt uit: ‘De beweging van de bolletjes vertelt ons wat er vervolgens met het DNA gebeurt. Als we een eiwit toevoegen dat het DNA compacter maakt, zoals ons histon-eiwit, dan zien we dat de bolletjes minder gaan bewegen. Als het DNA langer wordt, zul je juist meer beweging zien. Zo kun je indirect informatie opdoen over de functie van een eiwit.’
Al vroeg in de evolutie ontstaan
Het onderzoek leert ons meer over de werking van histonen in bacteriën, maar werpt ook nieuw licht op de vroege evolutie van deze cruciale eiwitten. Het feit dat een eenvoudige vorm van histonen al in sommige bacteriën voorkomt, suggereert dat ze al vroeg in de evolutie zijn ontstaan. ‘Het is lastig om dat precies te duiden’, aldus Dame. ‘Maar onze ontdekking toont aan dat bacteriële histonen mogelijk een vroege, basale vorm zijn van de complexere eiwitbollen die we vinden in eukaryoten en archaea. Zelfs eenvoudige vormen van leven gebruikten blijkbaar al verfijnde mechanismen om hun genen te beheren. Zo krijgen we stukje bij beetje een dieper begrip van fundamentele overeenkomsten en verschillen tussen organismen in onze evolutionaire stamboom.’
Wetenschappelijk artikel
Yimin Hu, Samuel Schwab, Silvia Deiss, Pedro Escudeiro, Thor van Heesch, Joe D Joiner, Jocelyne Vreede, Marcus D Hartmann, Andrei N Lupas, Birte Hernandez Alvarez, Vikram Alva, Remus T Dame, Bacterial histone HBb from Bdellovibrio bacteriovorus compacts DNA by bending, Nucleic Acids Research, 2024;, gkae485, https://doi.org/10.1093/nar/gkae485
